Genotipificar, es el proceso de determinación del genotipo de una variante en el ADN específico de un organismo biológico, mediante un procedimiento de laboratorio.
La genotipificación del virus del sarampión es muy importante para no seguir vacunado a las personas con vacunas con un genotipo que probablemente ya no sea responsable de las epidemias . Ahora están circulando los genotipos B3 y D8 , y estos virus no están tan neutralizados por los anticuerpos contra el genotipo de la vacuna (es decir, el genotipo A) como asi tambien por los anticuerpos generados contra las nuevas cepas.
El objetivo de la OMS era erradicar el sarampión en el Siglo XX, pero el sarampión sigue desafiando a los empesinados a erradicarla con la vacuna. Periódicamente se informan brotes epidémicos y algunos de ellos causados por la vacuna propiamente dicha.
Los brotes epidémicos afectan tanto a los no vacunados como a los individuos vacunados, es decir, personas inmunizadas contra
sarampión.
Un clon, la cepa B3, que era inicialmente importado de África y luego del este Europa a Europa central y los EE.UU., está involucrado en la reaparición mundial del sarampión, además al genotipo D8 . Los trabajadores de la salud son los más afectados por brotes esporádicos de sarampión en Europa.
Sarampión básico
Se ha definido como una enfermedad relacionada con los viajes y
se propaga en escuelas y hospitales.
En comparación con la distribución en 2011, el genotipo B3 ahora se distribuye globalmente. Se ha informado como el más transmisible que otros genotipos.
Por comparar la virulencia de los genotipos de sarampión en un modelo macaco, El Mubarak y colegas describió el genotipo B3 (de Sudán) como más patógeno que el genotipo europeo C2 (del Países Bajos), que era endémica en Europa antes del 2007.
La primera campaña de inmunización contra el sarampión comenzó en
la década de 1960, con una vacuna de una dosis. Luego, en la década de 1980, la OMS recomendó una segunda dosis de vacuna para aumentar
La cobertura inmune de los niños que no respondierona la primera dosis.
Los Centros para el Control y la Prevención de Enfermedades de EE. UU.
propuso una tercera dosis de sarampión, paperas y rubéola . En Europa, surge la pregunta de si incluso se debe proponer una cuarta dosis a los trabajadores de la salud, y si volver a vacunar a ellos cada 5 años podría garantizar una vacunación suficiente cobertura contra nuevas cepas.
Es bien sabido cómo los virus tienen la capacidad de mutar, especialmente en condiciones de presión selectiva (es decir, exactamente lo que estamos haciendo con las campañas de vacunación), y es por esta razón hablar de erradicación del «sarampión» es una contradicción. ¿Por qué estamos vacunando a millones de personas contra un genotipo que probablemente ya no sea responsable de las epidemias? Pero sobre todo: ¿por qué no procedemos a la genotipación en todos los sujetos diagnosticados con la enfermedad? ¿El propósito es la inmunización y la protección de la salud, o están vacunando a todos, independientemente de la necesidad o efectividad de esta intervención?
Genotipificación molecular de las cepas del virus sarampión
Condori-Yujra F, Jimenez-Vasquez V, Enríquez-Alva G, Gordillo-Vilchez S, Cabezudo-Pillpe N. Genotipificación molecular de las cepas del virus sarampión en el Perú, 2018. An Fac med . 7 jun. 2018 [citado 5ene.2020];79(1):94-5. https://revistasinvestigacion.unmsm.edu.pe/index.php/anales/article/view/14599 DOI: https://doi.org/10.15381/anales.v79i1.14599
En el año 2017, cuatro países de la región de las Américas notificaron casos confirmados de sarampión, a saber:
- Argentina (3 casos),
- Canadá (45 casos),
- los Estados Unidos de América (120 casos) y
- la República Bolivariana de Venezuela (727 casos).
En los primeros meses de 2018, los siguientes 9 países han notificado casos confirmados:
- Antigua y Barbuda (1 caso),
- Brasil (14 casos),
- Canadá (4 casos),
- Colombia (1),
- Estados Unidos de América (13 casos),
- Guatemala (1 caso), México (4 casos),
- Perú (2 casos) y
- Venezuela (159 casos)(1).
La epidemiología molecular de los virus del sarampión es de vital importancia en la vigilancia de cepas de sarampión en la circulación mundial, especialmente durante los brotes. La genotipificación del virus del sarampión puede jugar un papel importante en el seguimiento de las vías de transmisión durante la investigación del brote, por lo que, los resultados de genotipificación ayudan a confirmar, refutar o detectar las conexiones entre casos. Si dos casos tienen genotipos coincidentes, estos pueden estar conectados incluso si dicho nexo no es evidente (2).De acuerdo con el análisis de laboratorio realizado por la Fundación Oswaldo Cruz (Fio-cruz/RJ), el genotipo identificado en todos los casos confirmados es el D8. Este genotipo es idéntico al identificado en Venezuela en 2017 (3). En nuestro país fueron confirmados dos casos en 2018 (Juliaca y Callao), de estos se obtuvieron aislamientos virales con el objetivo de caracterizarlos molecularmente.
Las muestras de hisopado nasal y faríngeo, procedentes de Callao (paciente de 46 años) y Juliaca (paciente de 16 años), ambos con ELISA IgM positivo y PCR en tiempo real positivo, fueron inoculadas en líneas celulares Vero, tras lo cual se realizaron pasajes cada 7 días.
Las muestras fueron de pacientes con síntomas clínicos de sarampión recepcionadas en el Instituto Nacional de Salud (INS) para diagnóstico durante los meses de febrero y marzo del presente año. Se extrajo el ARN viral de las muestras seleccionadas utilizando un Kit de extracción QIAamp Viral RNA Mini, el cual fue almacenado a -40°C hasta su uso. Se realizó una PCR por transcripción reversa (RT-PCR) del gen de Nucleoproteína viral de sarampión (NP), los productos fueron revelados en un gel de agarosa al 2%, con ello se obtuvo un fragmento de 450pb. Los amplicones fueron posteriormente purificados con el método Exosap y secuenciados con el método de Sanger, en un analizador genético Applied Biosystems 3500. Las secuencias forward y reverse de cada amplicón fueron editadas y ensambladas con el software SeqTrace versión 0.9.0 y luego contrastadas con secuencias de nucleo-proteína de sarampión depositadas en la base de datos GeneBank (4). Se contó con secuencias de todos los genotipos conocidos, pero con mayor énfasis en secuencias del genotipo D8. Las secuencias fueron alineadas con el programa MAFFT versión 7. Se realizó una reconstrucción filogenética con la metodología de máxima verosimiltud (MV) con el programa RaXML version 8.0.0 con una búsqueda inicial de 50 árboles. Obtenido el árbol MV, se identificaron en él las secuencias más próximas a las muestras peruanas y se procedió con una última reconstrucción con la metodología Neighbor-Joining (NJ), con un número menor sesecuencias, opción de pairwise deletion y 10000 repeticiones bootstrap, esto último para estimar los soportes estadísticos de los nodos del árbol. (Figura 1).
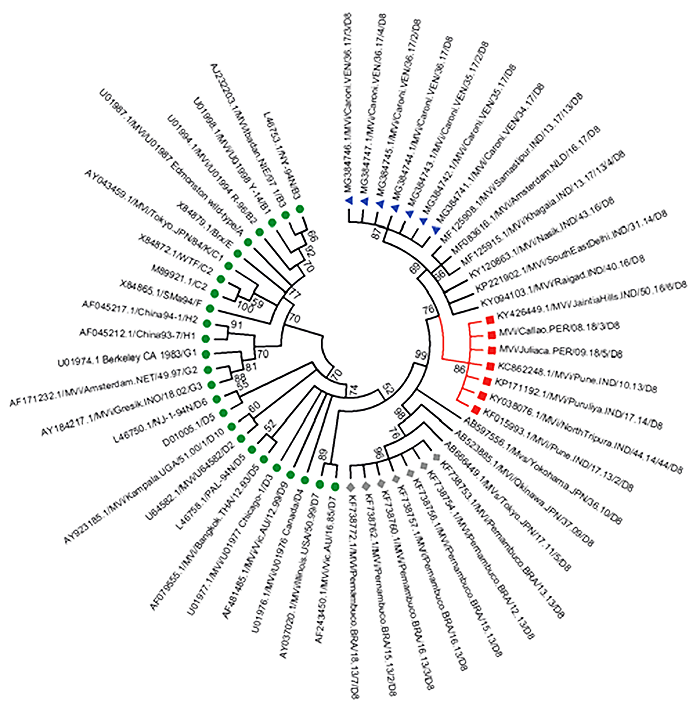
El árbol inicial obtenido con la metodología de máxima verosimilitud logró agrupar a las muestras peruanas en un clado conformado por muestras provenientes de la India, con aquella información se obtuvo un árbol más pequeño que permitió una mejor visualización de los vínculos entre las muestras de Perú, Venezuela, Brasil e India. Este último árbol estimado con la metodología NJ permitió corroborar el vínculo entre las muestras de Perú y la India, pertenecientes al genotipo D8, con un soporte estadístico bootstrap de 86%.”
Los análisis realizados, por tanto, permitieron caracterizar genotípicamente a las muestras de Perú como perteneciente al genotipo D8 y vinculada con brotes de la India. India justamente de donde Argentina esta comprando las vacunas.
- 1. Boletín Semanal de Sarampión/Rubéola: Organización Panamericana de la Salud. OPS/OMS. Vol.24, n.9 marzo de 2018. Disponible en: http://bit.ly/2HeaPZt.
- 2. World Health Organization: Standardization of the nomenclature for describing the genetic characteristics of wild-type measles viruses WHO/Wkly. Epi Rec. 1998; 73, 265-269.
- 3. World Health Organization, Regional Office for Europe. Press Release: Europe observes a 4-fold increase in measles cases in 2017 compared to previous year. Copenhagen. Acceso 19 febrero 2018. Disponible en: http://bit.ly/2ETCTnd
- 4. GeneBank del Centro Nacional de Información Biotecnológica de los Estados Unidos. Disponible en: https://www.ncbi.nlm.nih.gov/genbank/.
¿Qué es un genotipo?
Como su nombre indica, el genotipo de una persona se refiere a los tipos de genes que tiene para un rasgo hereditario particular. Los genotipos determinan qué características expresará un individuo, por ejemplo: si tiene pecas o no, si son intolerantes a la lactosa o si sus ojos serán azules, marrones u otro color.
¿Cómo surgen los genotipos?
Los genes se encuentran en los cromosomas , esas estructuras de ADN compactas en el núcleo celular. En los organismos de reproducción sexual, los cromosomas vienen en pares, uno de la madre y otro del padre. Por ejemplo, cada persona tendrá dos ‘Cromosomas 1’ y dos ‘Cromosomas 2’.
Los cromosomas en un par (excepto los cromosomas sexuales) se denominan cromosomas homólogos porque contienen los mismos genes.
Siempre hay dos copias de cada gen, una de cada padre. Sin embargo, un gen puede tener diferentes versiones, llamadas alelos. Los alelos son varias versiones de un gen. La combinación de alelos heredados de los padres es lo que da lugar a los genotipos.
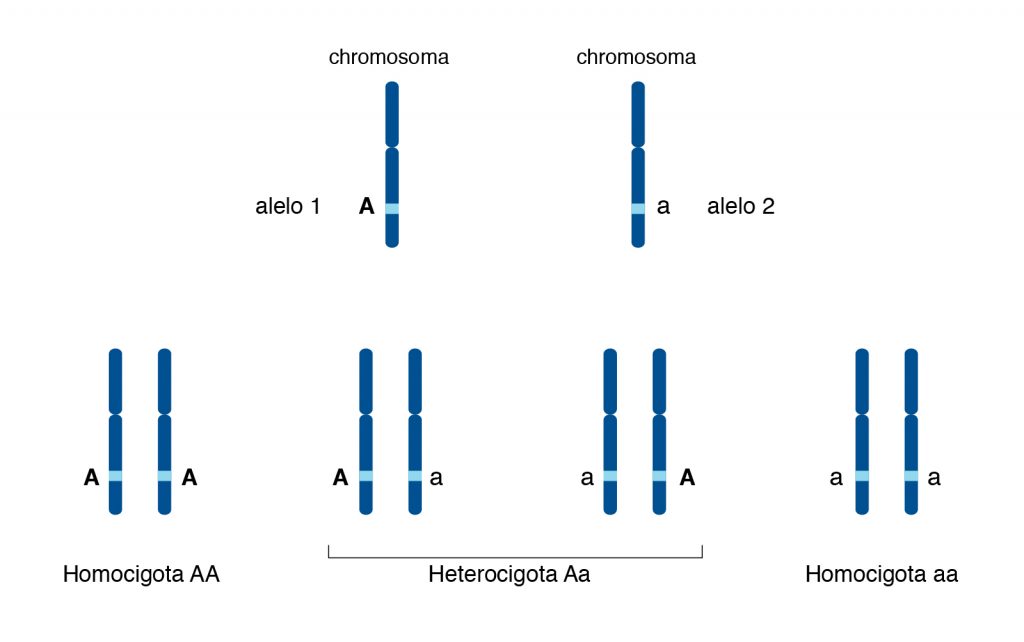
Ejemplos de alelos y genotipos
Para comprender mejor cómo funcionan los alelos y los genotipos, echemos un vistazo al gen que determina si una persona es intolerante a la lactosa, que se encuentra en el cromosoma 2. Este gen contiene la receta para que el cuerpo produzca lactasa, la enzima que descompone la lactosa. azúcar en la leche.
El alelo para la enzima lactasa está representado por una letra mayúscula porque es dominante sobre la producción defectuosa de lactasa. El alelo ‘más débil’ se conoce como recesivo y usa una letra ‘minúscula’. Entonces, siempre que la persona tenga ambas versiones del gen, en este caso Ll, la capacidad de producir lactasa, siendo el alelo dominante, será la que se exprese. Para que alguien sea intolerante a la lactosa, tiene que haber heredado dos alelos recesivos ‘l’, uno de cada padre.
 CienciaySaludNatural.com
CienciaySaludNatural.com